A crise global de saúde pública deflagrada pela emergência das superbactérias — como são popularmente conhecidas as bactérias resistentes a múltiplas classes de antibióticos, que causam infecções de difícil tratamento, sobretudo no ambiente hospitalar — tem ganhado relevância crescente, não apenas pelos alarmantes índices de morbidade e mortalidade associados a essas infecções, cujas opções de terapia são extremamente limitadas, ou, em alguns casos, inexistentes, mas também por seus impactos na economia a curto e longo prazo. Cerca de 700 mil óbitos ocorrem anualmente por infecções causadas por bactérias resistentes aos antibióticos, e as previsões para as próximas décadas não são otimistas. A estimativa projetada por um estudo encomendado pelo Reino Unido é de que, até 2050, esse número chegue a 10 milhões, superando a quantidade de óbitos causados por câncer. Considerando que essa estimativa foi realizada antes da pandemia de covid-19, esses números podem ser alcançados ainda mais rapidamente, tendo em vista que a intensificação do uso de antibióticos nesse período, como a azitromicina, pode favorecer a seleção, e consequentemente, a disseminação de bactérias resistentes a essa e outras drogas utilizadas para o tratamento de diversos tipos de infecções.
A demanda de soluções para o enfrentamento dessa grave crise, que parece nos conduzir a uma era pós-antibiótica, tem estimulado o desenvolvimento de diversas pesquisas, cujos objetivos vão desde o estudo de medidas profiláticas, como a pesquisa de alvos moleculares para a produção de vacinas, até as mais diferentes propostas de intervenção terapêutica, que compreendem o desenvolvimento de novas drogas e de metodologias alternativas, como a fagoterapia e antimicrobianos baseados em estratégias de manipulação genética, sobretudo em CRISPR-Cas9.
A revolucionária tecnologia de edição genômica CRISPR-Cas9, que rendeu o prêmio Nobel de química, em 2020, às cientistas Jennifer A. Doudna e Emmanuelle Charpentier, possui elevado potencial para as mais diversas finalidades biotecnológicas na manipulação de genomas de eucariotos e procariotos. Antes de mais nada, para que possamos entender como essa tecnologia funciona, é importante saber que ela não foi simplesmente inventada, mas sim descoberta. Isto é, originalmente o CRISPR é uma região especial dos genomas de parte dos procariotos, bactérias ou arqueias, caracterizada por repetições nucleotídicas palindrômicas intercaladas por espaçadores, que são sequências provenientes de DNA exógeno, o que explica a sigla que vem do inglês Clustered Regularly Insterspaced Short Palindromic Repeats (CRISPR), em tradução literal: Repetições Palindrômicas Curtas Agrupadas e Regularmente Interespaçadas.
O papel original do CRISPR no genoma desses microrganismos está associado à proteção contra invasores, como os vírus bacteriófagos, que podem infectar bactérias e induzi-las à morte após dominar o seu genoma em benefício próprio. Dessa forma, as bactérias dotadas de CRISPR podem adquirir trechos do DNA desses invasores, que, ao serem incorporados no locus CRISPR como espaçadores, conferem um tipo de memória imunológica, e, no caso de uma futura infecção pelo mesmo agente, sua degradação será promovida de forma articulada pelas proteínas efetoras Cas (do inglês CRISPR associated), como a Cas9, cuja atividade é orientada por um RNA-guia de dupla-fita formado a partir da transcrição do CRISPR. Assim, a reprodução do material viral no interior da célula bacteriana é neutralizada através da clivagem executada pela Cas9, bloqueando a infecção.
A alta especificidade da Cas9 na clivagem de ácidos nucleicos e a sua ação guiada por RNA permitem a construção de complexos adaptáveis para a promoção de cortes de alta precisão no DNA, de forma menos laboriosa e complexa e mais acessível, superando limitações associadas às técnicas anteriormente disponíveis. A ferramenta CRISPR-Cas9 conta com dois componentes principais para a edição genômica: o locus CRISPR, que é transcrito em pequenos RNAs cognatos aos trechos específicos a serem editados no genoma, e a proteína Cas9, que corta o DNA com a sinalização prévia do RNA-guia. No contexto da resistência aos antibióticos, essa metodologia pode ser empregada para o nocauteamento de genes associados á resistência aos fármacos utilizados na clínica, promovendo a ressensibilização de cepas bacterianas resistentes e a manutenção da eficácia da antibioticoterapia convencional (Figura).
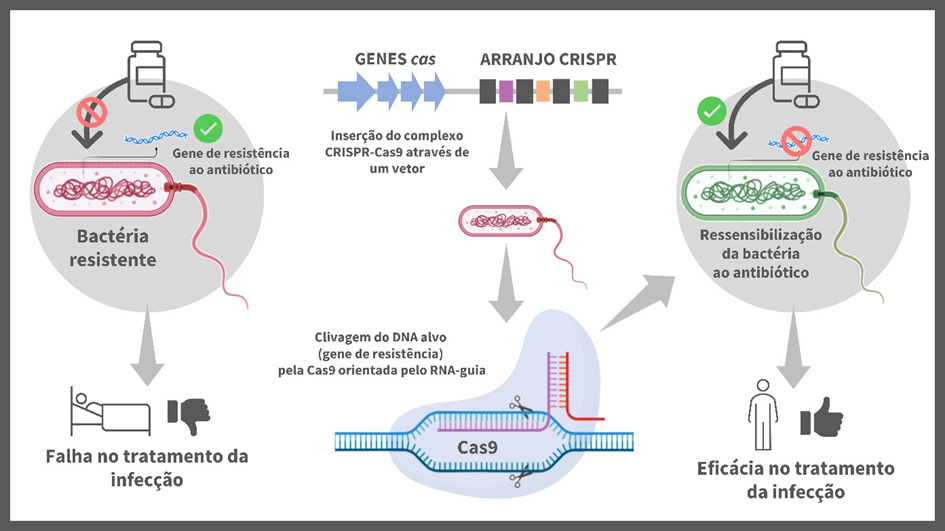
Essa metodologia tem sido aplicada em experimentos com diferentes espécies bacterianas, revelando resultados satisfatórios que sustentam a aplicação de abordagens baseadas em CRISPR-Cas9 como alternativas promissoras para o controle da disseminação da resistência aos antibióticos. Um dos maiores desafios para a viabilização de procedimentos baseados nesse mecanismo é a entrega dos complexos CRISPR-Cas9 programados para as células-alvo, levando em consideração as peculiaridades dos envoltórios de bactérias Gram-positivas e Gram-negativas e a necessidade de transposição de complexos proteína-RNA de, aproximadamente, 160 kDa. Para isso, diferentes estratégias têm sido investigadas: a construção de phagemids, que são plasmídeos portadores do complexo desenhados para serem empacotados no capsídeo de bacteriófagos específicos para as bactérias alvo; nanocomplexos de CRISPR; mecanismos de conjugação e outros.
Um estudo colaborativo de pesquisadores da Universidade do Texas em Dallas e da Universidade do Colorado, nos Estados Unidos, comprovou que cepas doadoras de plasmídeos vetores dos complexos CRISPR-Cas9 — nesse caso, desenhados para clivagem de genes de resistência na espécie Enterococcus faecalis —foram capazes de contribuir para a depleção da resistência mediada pela expressão dos respectivos genes alvo do CRISPR in vivo no intestino de murinos. Tais resultados indicam que a produção de probióticos com cepas doadoras desses complexos CRISPR-Cas9 pode ser útil para a erradicação de cepas multirresistentes específicas, constituindo grande potencial para descolonização de pacientes hospitalizados, os quais são altamente vulneráveis à aquisição de infecções oportunistas no ambiente hospitalar. Outros trabalhos seguindo a mesma abordagem de degradação de genes associados à resistência têm sido realizados em outras espécies de importância na etiologia de infecções hospitalares, como Staphylococcus aureus e Escherichia coli, sendo também bem-sucedidos na ressensibilização de cepas aos antibióticos in vitro e in vivo em modelos animais.
A tecnologia CRISPR-Cas9 pode ser uma ferramenta muito útil no controle da disseminação da resistência aos antibióticos e no combate a infecções causadas por cepas resistentes a múltiplas drogas, sendo um mecanismo preciso e capaz de discriminar microrganismos benéficos e patogênicos, o que denota uma estratégia valiosa na administração dessa crise, ao passo que contorna as limitações de custos de pesquisa e de tempo inerentes ao desenvolvimento e implementação de novas classes de antimicrobianos. Os esforços colaborativos de cientistas ao redor do globo, na última década, caracterizam o CRISPR como um promissor campo de investigação, já tendo sido alcançados avanços notáveis na aplicabilidade desses sistemas. Mas ainda existe espaço para aperfeiçoamentos e para a maior compreensão de algumas lacunas de conhecimento, sobretudo em relação às técnicas de entrega dos complexos CRISPR-Cas9 e sobre os impactos ecológicos em comunidades microbianas complexas, a longo prazo.
Em busca de garantir a segurança e aprimorar o controle da utilização do sistema CRISPR-Cas9, são necessários estudos que visem a otimização da técnica, sobretudo na investigação de possíveis limitações associadas ao seu funcionamento. Mecanismos como proteínas anti-CRISPR, também denominadas Acr, podem trazer uma boa perspectiva quanto ao aumento da probabilidade de êxito e da precisão das aplicações de CRISPR-Cas9. Essas proteínas inativam o sistema CRISPR-Cas9 através de mecanismos variados, por exemplo, bloqueando os domínios endonuclease da Cas9, o que permite a evasão do seu mecanismo de ação.
As proteínas anti-CRISPR foram descobertas, inicialmente, em bacteriófagos e a sua aplicação na edição de genomas bacterianos, bem como em células humanas modificadas, vem sendo testada para o aperfeiçoamento dessa tecnologia, sendo aplicada no desligamento da Cas9 e, consequentemente, na redução de efeitos off-target e da citotoxicidade resultante da atividade excessiva da nuclease nos tecidos. Outros estudos também apresentaram estratégias artificiais de regulação das proteínas anti-CRISPR, como a adição de um domínio fotossensível na proteína, que permite que a atividade anti-CRISPR seja controlada por luz azul. Dessa forma, a regulação de proteínas Acr por promotores induzíveis, luz ou pequenas moléculas, pode contribuir para um controle eficiente, dinâmico e espacial da atividade CRISPR-Cas. O pequeno tamanho das proteínas Acr (~ 50-200 aminoácidos), que implica na possibilidade de entrega in vivo por meio de vetores AAV, e a capacidade de desligamento da atividade da Cas9 oferecida por essas moléculas, representam características interessantes a serem exploradas em direção ao aperfeiçoamento das terapias baseadas em CRISPR-Cas9.
Para mais informações sobre o tema, recomendamos a leitura dos artigos e documentos disponíveis nos links abaixo:
- https://amr-review.org/sites/default/files/160518_Final%20paper_with%20cover.pdf
- https://www.nature.com/articles/nm.4465
- https://mbio.asm.org/content/7/3/e00428-16
- https://doi.org/101128/CMR.00066-18
- https://doi.org/10.1016/j.tibtech.2017.10.021
- https://doi.org/10.1016/j.jmb.2018.09.013
- https://doi.org/10.1186/s12941-019-0317-x
- https://doi.org/10.1128/AAC.01454-19.
- http://dx.doi.org/10.1016/j.drup.2016.11.001
- https://www.nature.com/articles/nbt.3043
- https://doi.org/10.1016/j.omtm.2018.04.008
- https://doi.org/10.1038/nature22377
- https://doi.org/10.1126/sciadv.1701620
- https://doi.org/10.1038/s41592-018-0178-9
- https://doi.org/10.1038/nature11723
Autoria: Vitor Luis Macena Leite, Adriana Rocha Faria & Lúcia Martins Teixeira



0 comentário