Desde os primeiros registros de enterococos resistentes à vancomicina (VRE) na década de 1980, estudos relatam os impactos negativos à saúde e economia causados por esses microrganismos, sendo fundamental a sua detecção precisa para a prevenção de surtos e a aplicação da terapia adequada. A resistência adquirida à vancomicina é conferida por uma série de variantes do operon van, sendo o operon vanA mais comum e, portanto, clinicamente mais relevante. A resistência à vancomicina codificada por vanA é mediada pelo grupo de genes vanR, vanS, vanH, vanA, vanX, vanY e vanZ que geralmente são transportados no transposon Tn1546. A expressão desses genes leva à substituição do resíduo D-Ala C-terminal por D-Lac durante a síntese da parede celular, modificando assim o alvo de ligação da vancomicina. O transposon está frequentemente contido em plasmídeos, facilitando a disseminação, sobretudo, entre amostras clínicas de Enterococcus faecium e Enterococcus faecalis.
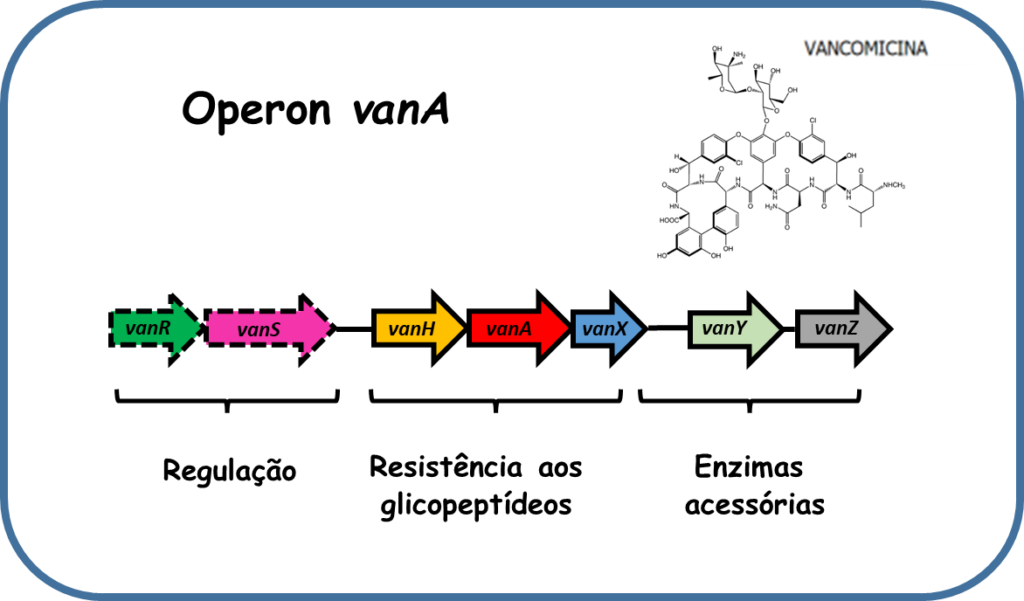
O termo enterococos variáveis à vancomicina” (VVE, do inglês vancomycin-variable enterococci) vem sendo usado para caracterizar amostras suscetíveis à vancomicina contendo o genótipo vanA (VVE-S), e capazes de reverter para um fenótipo de resistência após a exposição à vancomicina (VVE-R). Uma vez que a expressão dos genes de resistência à vancomicina está desligada nos VVE-S, eles não são facilmente detectados por meio de métodos fenotípicos tradicionais. No entanto, a exposição durante o tratamento com glicopeptídeos, ao longo de dias, promove a expressão de genes vanHAX, e o consequente desenvolvimento de resistência. Assim, os VVE são responsáveis por falhas terapêuticas, e podem ser considerados uma ameaça emergente. Os genes vanR (um regulador da resposta citoplasmática) e vanS (sensor de glicopeptídeo) compõem um sistema de transdução de sinal de dois componentes que controla a expressão dos genes que medeiam a resistência aos glicopeptídeos. Na presença de vancomicina, a proteína VanR é fosforilada por VanS para promover a transcrição do operon. A ausência total ou ineficiência deste sistema pode ser responsável pela detecção de VVE.
O fenômeno foi bem documentado por Coburn et al., (2014) durante o relato do caso de um paciente no Canadá. Os autores relataram o surgimento de resistência à vancomicina (CIM >256 µg/mL no dia 22 do curso terapêutico) em um paciente colonizado por E. faecium inicialmente suscetível à vancomicina (CIM 1 µg/mL no dia 0). Durante o período de internação do paciente, a coleta de swabs retais nos dias 10, 12, 22 e 24, fluido ascítico nos dias 2 e 14, e urina no dia 10, permitiram o isolamento de E. faecium carreando o gene vanA. Entretanto, a resistência à vancomicina foi detectada apenas a partir do 22º dia de internação, após o início do tratamento com vancomicina no dia 12. Os perfis de fragmentação obtidos após eletroforese em campo pulsado (PFGE) de todas as amostras isoladas foram indistinguíveis. Os plasmídeos das amostras isoladas nos dias 2 e 24 foram sequenciados e, em ambas, tanto na sensível quanto na resistente à vancomicina, os plasmídeos continham vanHAXYZ, mas não vanRS. Embora sequências de inserção sejam causas frequentes de alterações estruturais em vanRS e vanHAXYZ, os autores relatam que o sequenciamento do genoma completo não revelou a presença de inserções cromossômicas adicionais, nem a presença de vanRS, homólogos de vanRS ou mesmo outro operon de resistência à vancomicina.
Embora a incidência de VVE-S seja desconhecida, já foram documentados surtos no Canadá, Dinamarca e Noruega, além de casos esporádicos na Coreia e na Bélgica. As amostras VVE relatadas foram identificadas, sobretudo, como E. faecium, com ausência ou deficiência de vanRS. Assim, a presença de VVE após a exposição terapêutica representa um risco, uma vez que os métodos tradicionais de cultivo seguidos por testes de suscetibilidade não permitem a identificação dessas amostras, sendo necessária a adoção de métodos fenotípicos e moleculares, em conjunto.
Para mais informações sobre o tema, recomendamos a leitura dos artigos disponíveis nos links abaixo:
https://pubmed.ncbi.nlm.nih.gov/24523476/
https://pubmed.ncbi.nlm.nih.gov/32340989/
https://pubmed.ncbi.nlm.nih.gov/24523464/
https://pubmed.ncbi.nlm.nih.gov/33367710/
Autores: Andréa de Andrade Rangel de Freitas & Lúcia Martins Teixeira



0 comentário